一、基因编辑技术的更迭
1.同源重组(homologousrecombination,HR)
2.归巢内切酶(HomingEndonuclease,HEs)
3.第一代基因编辑技术—ZFNs技术
4.第二代基因编辑技术—TALENs技术
5.第三代基因编辑技术—CRISPR/Cas技术
6.CRISPR/Cas衍生的新一代靶向基因编辑
二、基因编辑技术的应用领域
1.疾病模型
2.靶向基因治疗
3.动植物育种
4.微生物设计
5.基因诊断与核酸检测
三、基因编辑技术的关键问题
1.脱靶效应
2.效率问题
3.运输问题
4.免疫排斥
5.副作用
6.伦理问题
四、基因编辑技术国内外相关政策与规划
1.中国
2.美国
3.科研共同体及其他国家
五、基因编辑技术的临床应用
1.罕见病
2.神经退行性疾病
3.年龄相关性黄斑衰退(AMD)
4.1型糖尿病(T1D)
5.艾滋病(HIV)
6.肿瘤
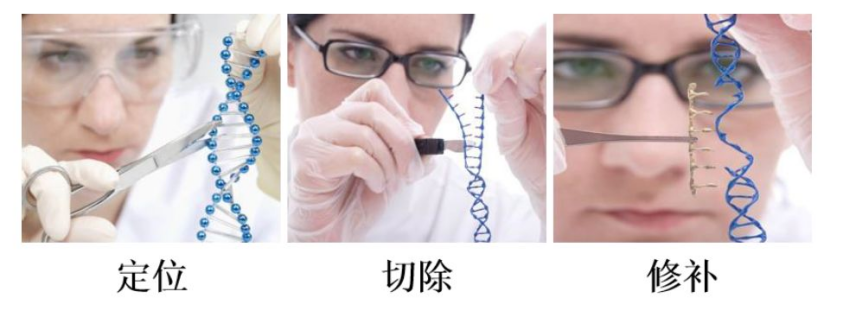
一般来说,基因编辑的过程可概括为三个步骤:定位、切除、修补。
来源:参考资料2
定位异常基因:在DNA上找到需要进行编辑的位置,即识别染色体上的DNA靶位点;
切除异常基因片段:定位到目标位置以后,通过内切酶将错误的基因片段切除;
修补恢复成正常基因:内切酶把错误的基因片段切除后,会在DNA上形成一段缺口,这时只需要把对应的正确基因片段插入到这个缺口中并与原DNA进行连接即可完成DNA的修复。
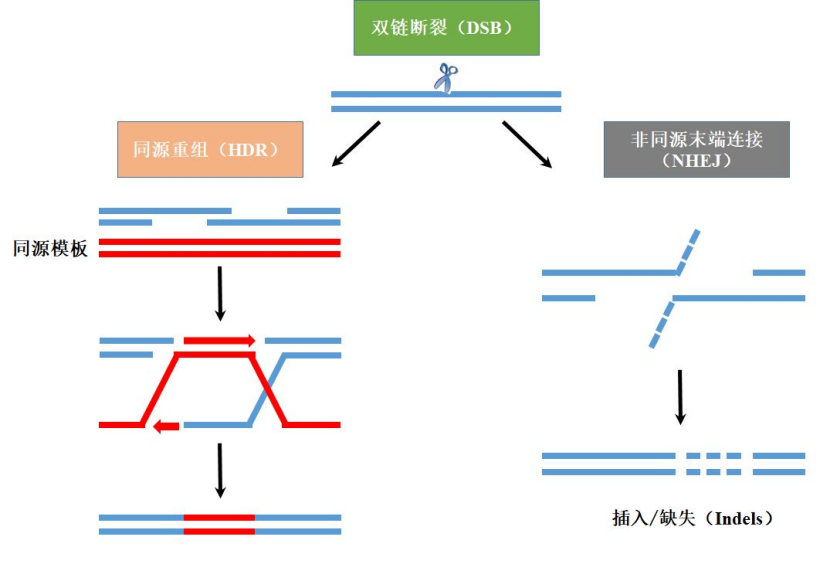
通常,修复缺口的机制包括:
①内源性的非同源末端连接(Non-homologous end-joining,NHEJ),这是细胞自身的随机修复机制,且不需要同源模板,可直接将DNA末端连接在一起,但NHEJ途径错配率较高,易造成DNA损伤位点碱基序列的改变(错配和丢失),从而导致遗传信息的丢失;
②同源重组修复(Homology-directed repair,HDR),即在有同源序列作为修复模板的条件下,外源的目的基因能通过同源重组整合入靶基因,降低了错误率;
③选择性末端修复(alternative end-joining,a-EJ)和单链退火修复(single strand annealing,SSA),作为辅助性的修复机制,a-EJ和SSA均需要更大幅度的末端单链切除,这也会导致遗传信息的丢失。
来源:参考资料3
作为生命科学发展迅速的重要研究领域,基因编辑技术的开发及应用使得生物体的遗传改造进入了前所未有的深度与广度。同时,该技术在基因功能研究、药物开发、疾病治疗等方面的广阔应用前景也使其成为了世界范围内竞争最为激烈的下一代核心生物技术之一。
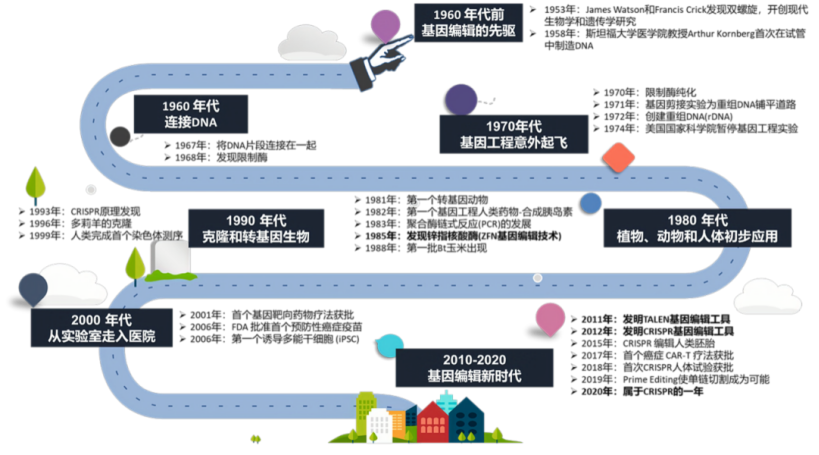
基因编辑技术的发展历程(来源:参考资料2)
一
基因编辑技术的更迭
近年来,随着对基因编辑技术的不断探索,多种高效新型核酸酶的发现使基因编辑技术发生了质的飞跃。
01
同源重组(homologous recombination,HR)
20世纪70年代,科学家在研究细菌如何防御噬菌体过程中发现限制性内切酶可以保护细菌免受噬菌体的侵害,这个发现具有里程碑式的意义,科学家发现可以对基因组进行编辑。20世纪80年代,Smithies等和Capecchi发现,可以通过同源重组将外源DNA整合到哺乳动物的基因组中,但这种方法有很大的局限性,不仅整合效率极低(整合效率取决于细胞的状态和类型),而且容易脱靶。
02
归巢内切酶(Homing Endonuclease,HEs)
20世纪80年代末期,Rudin等和Rouet等发现,在靶标位置引入双链断裂的DNA(double-strand break,DSB)会显著提高目的基因的整合效率。研究人员最早通过归巢核酸内切酶(也称大范围核酸内切酶)在基因组中引入特定的双链断裂DNA。归巢核酸内切酶能够识别14~40bp的DNA片段,识别之后绝大部分通过NHEJ途径修复。人们在自然界中已经找到数百种归巢核酸内切酶,使用最广泛的主要有3种:Ⅰ-SceⅠ、Ⅰ-CreⅠ和Ⅰ-DmoⅠ。
然而,通过归巢核酸内切酶进行基因编辑存在很大的制约性:
尽管有数百种的归巢核酸内切酶,每个都有自己独特的识别序列,但要找到适合靶向特定基因序列的归巢核酸内切酶的概率极低;
由于DNA双链断裂主要通过NHEJ途径修复,该修复机制不能引入外源的DNA模板,并且会在断裂位点随机删除或者插入DNA片段。

归巢核酸内切酶技术简图(来源:参考资料3)
03
第一代基因编辑技术—ZFNs技术
人工核酸酶技术的发展使人为定点诱导DSBs成为现实,其中锌指核酸酶技术(zinc finger nucleases,ZFNs)就是一个里程碑式的突破,也称为第一代基因编辑技术。
锌指蛋白(Zinc finger protein,ZFP)是一类通过Cys2-His2锌指结构域结合DNA的转录因子,由一个α螺旋和两个反向的β平行形成紧密的ββα结构,能特异性识别3个连续的碱基对。ZFNs是通过设计锌指结构域以及多个锌指蛋白的不同连接顺序实现特异性结合靶标基因和限制性核酸内切酶FokⅠ进行定点切割的技术。值得注意的是,FokⅠ核酸酶需要形成二聚体才可切割DNA产生DSB,为避免FokⅠ自身二聚化,将FokⅠ设计为异源二聚体,这样的设计也增强了ZFNs的结合特异性。
目前ZFNs可以通过HDR或NHEJ来修饰体细胞和多功能干细胞的基因组。该技术靶向结合效率高,但是蛋白设计复杂,费时费力,并且无法实现对任意靶基因的结合,也无法实现高通量的基因编辑。作为新型基因编辑工具,ZFNs从1996年问世,在2001年开始被陆续用于不同物种的基因编辑。
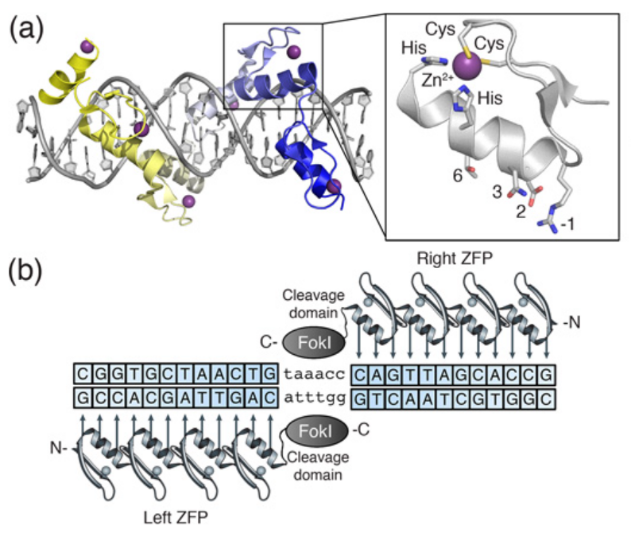
ZFNs结构图(来源:参考资料5)
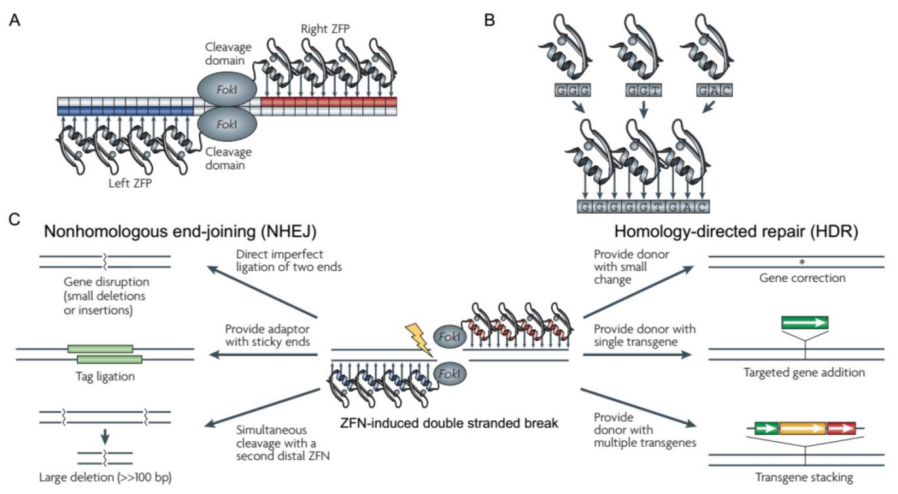
锌指蛋白基因编辑原理(来源:Nature Reviews Genetics)
然而该技术也有局限性:
可编辑的靶基因位点有限,有限的锌指蛋白类别只可识别有限的DNA序列;
ZFNs基因编辑的效率低,只有30%左右;
操作复杂,成本高,容易脱靶。
一直以来,科研人员都在不断改进ZFNs技术,2014年,Tebas等通过ZFNs编辑艾滋病毒(HIV)感染者自体CD4 T细胞中的艾滋病毒入侵辅助受体CCR5来治疗艾滋病。但是,目前ZFNs技术存在专利封锁。ZFNs核心技术专利掌握在数位科学家手中,这些科学家陆续加入或专利授权给美国Sangamo,其获得了ZFNs设计、筛选、优化、实验室和临床应用相关的数个关键专利,将其垄断。目前,ZFNs技术自问世以来无大规模应用,且无突破性进展。
04
第二代基因编辑技术—TALENs技术
类转录激活因子(transcription activator-like effector,TALE)是植物病原菌黄单孢菌中的一种效应因子。2009年,Boch等和Moscou等发现,类转录激活因子蛋白可以识别DNA序列。2011年,第二代基因编辑技术——类转录激活因子效应物核酸酶TALENs诞生。
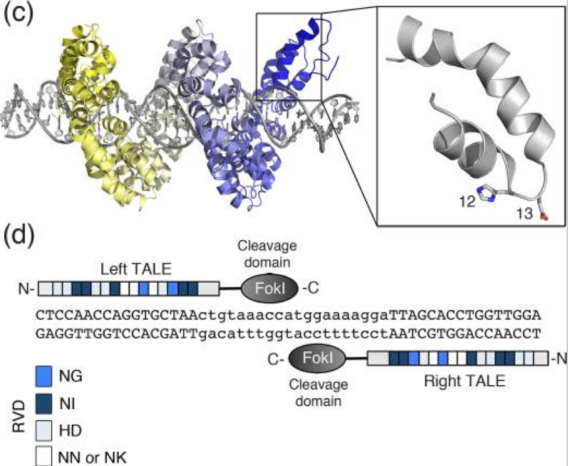
TALENs结构图(来源:参考资料5)
与ZFNs一样,TALENs在形式和功能上也是模块化的,由FokⅠ切割结构域和TALE蛋白DNA结合结构域融合组成,同样利用限制性核酸内切酶FokⅠ进行基因编辑。
TALE蛋白由33–35个氨基酸的重复单元组成,通过位于12位和13位的两个可变氨基酸残基识别并结合DNA序列,这两个位点的氨基酸残基称为重复可变的双氨基酸残基(repeat variable diresidue,RVD),每个重复单元能特异性识别一个碱基对。
TALENs的唯一靶向限制是对N端结构有5'T的要求,因此,通过构建不同的RVD和改变TALE的连接顺序,理论上可以使TALENs被用于几乎任何的序列。2011年TALENs技术开始用于人类细胞的基因编辑,如靶基因的敲除等。
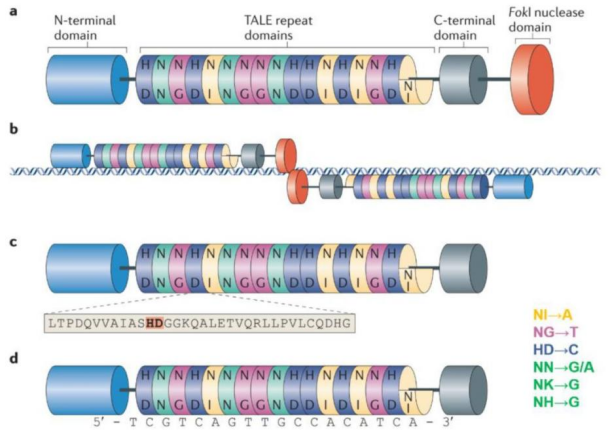
TALENs基因编辑原理(来源:Nature Reviews Molecular CellBiology)
与ZFNs相比,TALENs的毒性低,蛋白设计相对简单,是首个真正意义上的基因可编辑工具。但是TALENs重复序列更多,工作量巨大,此外,TALENs的体积比ZFNs大,这使得某些病毒传递系统的包装较困难,也无法用于高通量的编辑。
05
第三代基因编辑技术—CRISPR/Cas技术
5.1 CRISPR/Cas的发现
CRISPR/Cas(clustered regularly interspaced short palindromic repeats / CRISPR-associated proteins)技术又被称为第三代基因编辑技术,是基于原核生物(细菌和古生菌)一种免疫系统而开发的,是目前应用最为广泛的基因编辑工具。
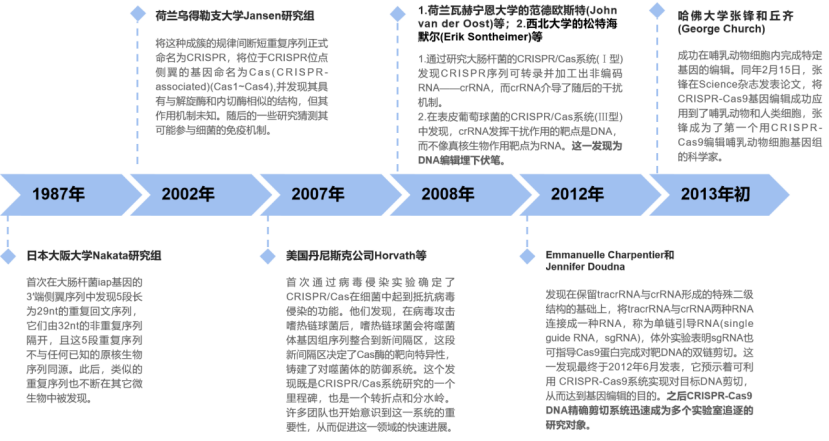
CRISPR/Cas发展史大事记(来源:公开资料,小编自制图)
在CRISPR/Cas的发展史中,值得注意的是,其实2010年,科学家们就已经对CRISPR/Cas系统的基本生物学作用和分子机制有了较为清晰的理解,并将其应用于减少细菌噬菌体感染和细菌进化分析等,但当时CRISPR/Cas系统的应用范围极为有限。主要原因在于当时已研究的两种类型(Ⅰ型和Ⅲ型)都过于复杂,因此,寻找更为简单的体系成为了一个重要方向。

crRNA-tracrRNA改造为sgRNA示意图(A:天然化脓性链球菌内的“靶向DNA-crRNA-tracrRNA-SpCas9”复合体;B:将crRNA和tracrRNA改造为sgRNA后的模式图。来源:参考资料6)
5.2 CRISPR/Cas的作用机制
经过多年的研究,人们对CRISPR/Cas系统的作用机制有了相对清晰的了解。以CRISPR-Cas9为例,细菌对外来病毒的入侵分为3步:
(1)病毒入侵时,CRISPR/Cas系统将病毒的DNA切成短片段,并插入重复序列之间,作为“记忆”储存;
(2)同种病毒再次入侵时,CRISPR阵列及Cas9基因转录,Cas9翻译为蛋白,转录出的tracrRNA与pre-crRNA互补配对,经过内源核糖核酸酶(RNase)加工成熟,最后形成Cas9-crRNA-tracrRNA的三聚体;
(3)在crRNA与病毒DNA互补配对之前,Cas9需要与特定的PAM序列结合以区别病毒和自身基因组,Cas识别并结合PAM后将DNA双链解旋,crRNA在PAM上游与目标序列互补配对。在PAM和靶点序列均匹配时,Cas9构象发生改变,其双链内切酶的活性被激活,在PAM上游的特定位置将病毒的双链DNA切断。

CRISPR的来历(来源 :https://research.sinica.edu.tw/gene-editing-crispr-lin-chia-hung/)
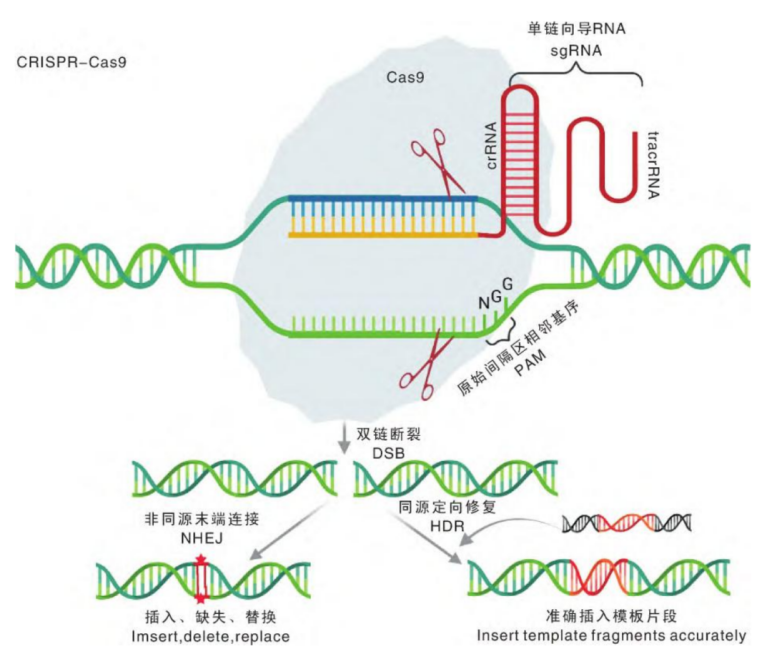
CRISPR-Cas9基因编辑原理(来源:参考资料8)

来源:参考资料4
此外,CRISPR/Cas系统还具有基因编辑效率更高、操作成本低等多方面的优势。然而,CRISPR技术出现时间还太短,科学家还无法充分预估其潜在风险。相比CRISPR技术,TALENs的脱靶效应更低,更加稳定。
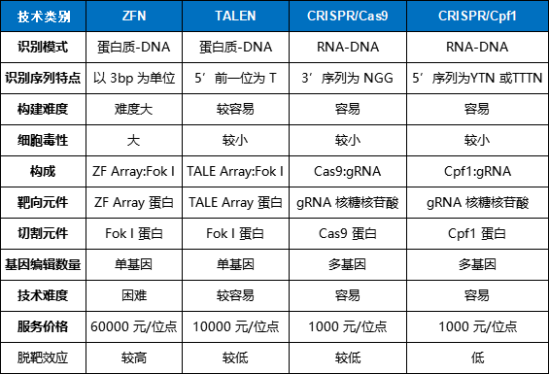
基因编辑技术参数对比(来源:参考资料2)
06
CRISPR/Cas衍生的新一代靶向基因编辑
6.1 碱基编辑技术(BE)
2016年和2017年,刘如谦实验室分别将胞嘧啶脱氨酶或腺苷脱氨酶与Cas9结合,从而开创性地建立了新一代基因编辑系统——碱基编辑器(Base Editor,BE)。BE是在CRISPR-Cas9技术的基础上设计的,由sgRNA和融合蛋白组成。其中,融合蛋白为经过改造的Cas9、胞嘧啶脱氨酶和尿嘧啶糖基化酶抑制子的复合体。
BE系统包含了胞嘧啶碱基编辑器(Cytidine Base Editors,CBE)和腺嘌呤碱基编辑器(Adenine Base Editor,ABE),可以在不需要模板DNA和双链DNA断裂的情况下分别实现胞嘧啶(C)与胸腺嘧啶(T)、腺嘌呤(A)与鸟嘌呤(G)核苷酸之间的转化。其中,ABE可实现A到G(或T到C)的转换,理论上可解决47%的基因致病突变导致的人类疾病。
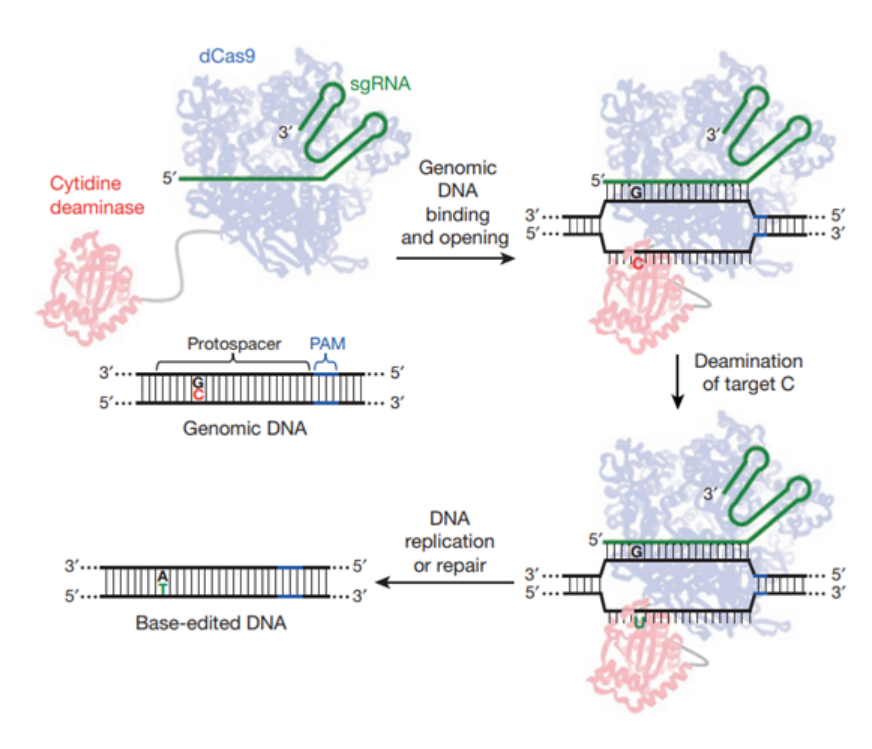
CBE技术原理(来源:参考资料10)
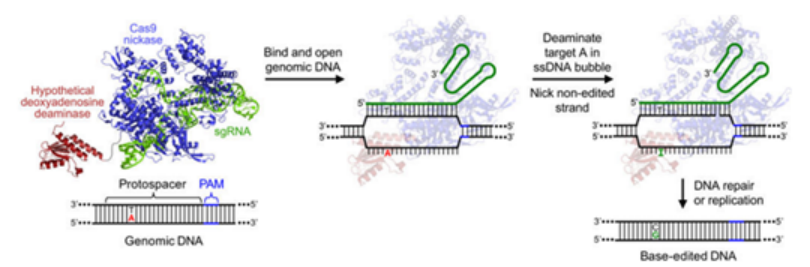
ABE技术原理(来源:参考资料10)
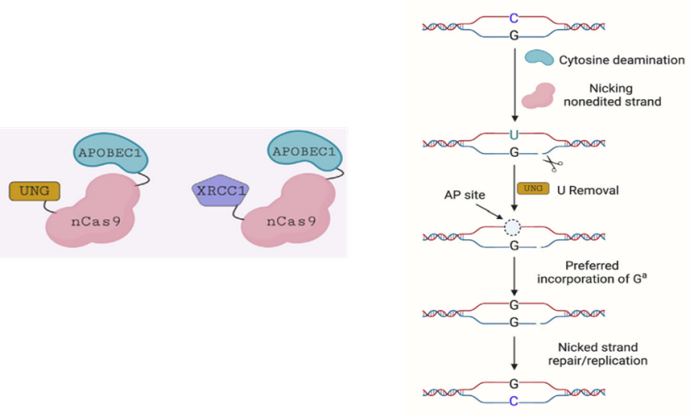
GBE技术原理(来源:参考资料8)
除上述以外,近些年来,研究人员通过将胞嘧啶脱氨酶、腺嘌呤脱氨酶和Cas9切口酶相融合,开发出了腺嘌呤和胞嘧啶双碱基编辑器(ACBE),这一双碱基编辑器可以在同一靶标位点实现C-T和A-G转化,研究表明ACBE的C-T编辑效率与CBE相当,对A-G编辑效率则略有降低;但总体来说该双碱基编辑器效率高于CBE+ABE。
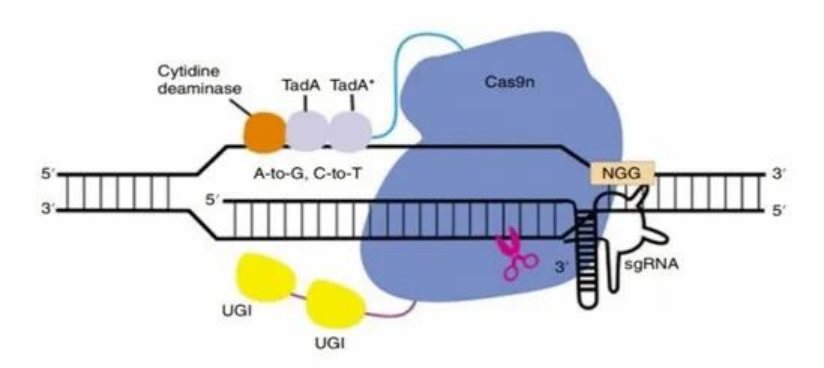
ACBE技术原理(来源:参考资料8)
另外,就在去年,赖良学课题组通过将GBE和ABE融合构建出了AGBE编辑器,由于GBE除可实现C-to-G转换外,还存在C-to-T和C-to-A两种副产物。因此利用AGBE对C-to-G、C-to-T、C-to-A和A-to-G的编辑特性,可用其建立饱和突变群体,以通过高通量筛选验证多种基因突变模式如单核苷酸变异(SNV)的功能及相关表型。
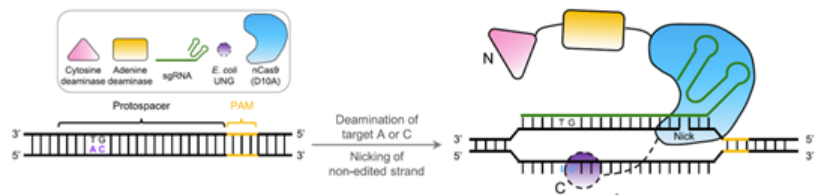
AGBE技术原理(来源:参考资料8)
BE的发现,在实现精确和高效的基因组编辑的同时将最大限度地减少不需要的副产物和与双链DNA断裂相关的细胞毒性。
但是,BE也存在着一定问题和局限性。一方面,现有的BE仅能完成特定的4种形式的碱基转换,缺乏完成精确可控的碱基颠换或者片段插入缺失等其他基因组编辑的能力;另一方面,BE往往仅能在向导RNA(guide RNA)识别区域中有限编辑窗口内发挥高效作用,编辑区域受到编辑器Cas蛋白PAM识别能力和编辑器窗口范围的双重限制,因此无法编辑基因组中一些重要位点。
此外,BE自身编辑的精确性还有待进一步优化。例如,编辑器脱氨酶,特别是CBE的胞嘧啶脱氨酶,存在序列非特异活性,可能在全基因组范围出现脱靶;同时当编辑窗口内存在多个可编辑碱基时,编辑器在编辑目标碱基时也会造成其他非靶向的碱基突变。
6.2 先导编辑(PE)
介于BE系统的脱靶现象,2019年,刘如谦团队报道了所开发的全新基因编辑技术——先导编辑(Prime Editor,PE,也称为引导编辑),不仅较好解决了脱靶的问题,而且可以实现全部12种可能的碱基转化。
PE系统以CRISPR-Cas9为基础,在sgRNA3’末端增加一段RNA序列,新获得的RNA被称作pegRNA,它不但能够结合想要进行编辑的特定DNA区域,还自带“修改模板”。除了可对单个碱基进行修改之外,这一系统还能够插入或删除特定的DNA序列。

目前,PE技术还不成熟,其可靠性有待进一步研究,而且与碱基编辑技术相似,使用逆转录酶和脱氨酶仍然存在安全隐患。
6.3 PASTE
不依赖DNA修复可编程多重整合大片段仍是现阶段基因组编辑的一个难题。目前的基因整合方法产生双链断裂,从而引发DNA损伤,并依赖于终末分化细胞中不活跃的DNA修复途径。PE尽管绕过了双链断裂,但仅限于修改或插入短序列。而新开发的PASTE系统正好解决了这一难题。
PASTE将丝氨酸整合酶与引导编辑结合,可在细胞中可编程高效多重整合大片段。丝氨酸整合酶包含AttP附着位点和靶向的AttB附着位点,通过将46bp的AttB附着位点设计在引导编辑的pegRNA中,将其插入到基因组中作为整合酶的靶点。这些蛋白可以以反式提供,也可以直接融合到Cas9蛋白中表达。同时递送含有AttP附着位点的环状双链DNA模板,目的基因可以在单次递送反应中直接整合到目标位点。
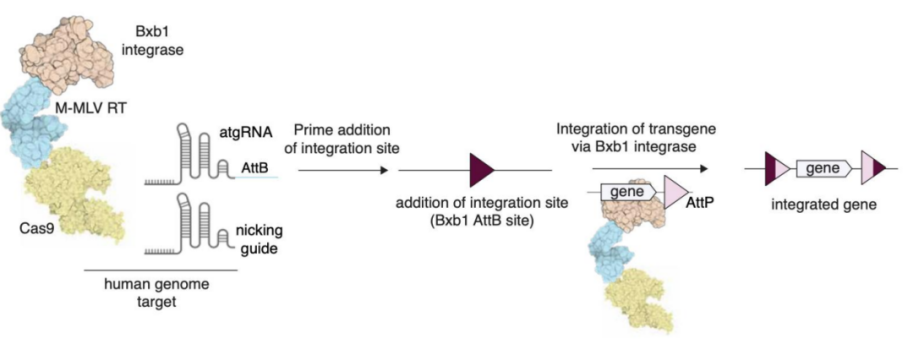
PASTE技术原理(来源:参考资料8)
目前,PASTE可在三种人类细胞系、原代T细胞和静止不分裂的原代人肝细胞的多个基因组位点上整合高达36kb的序列,效率在10-50%之间。PASTE的编辑效率与依赖于同源修复或非同源末端连接的整合相当或更好,在非分裂细胞中具有活性,且更少可检测到的脱靶事件。若应用于疾病治疗,PASTE可以用mRNA递送,可编程地直接插入腺相关病毒或腺病毒载体携带的DNA模板。
PASTE通过拖放式整合目的基因,扩展了基因组编辑能力,为研究、细胞工程和基因治疗提供了一个广泛适用的平台。






